基本数据管理
目标:
- 操作日期和缺失值
- 熟悉数据类型的转换
- 变量的创建和重编码
- 数据集的排序、合并与取子集
- 选入和丢失变量
案例:
R in action提供,用于研究男性和女性在领导各自企业上的不同
manager <- c(1,2,3,4,5)
date <- c("10/24/08","10/1/08","10/12/08",""5/1/09)
country <- c("US","US","UK","UK","UK")
gender <- c("M","F","F","M","F")
age <- c(32,45,25,39,99)
q1 <- c(5,3,3,3,2)
q2 <- c(4,5,5,3,2)
q3 <- c(5,2,5,4,1)
q4 <- c(5,5,5,NA,2)
q5 <- c(5,5,2,NA,1)
leadership <- data.frame(manager,date,country,gender,age,q1,q2,q3,q4,q5,stringAsFactors=FALSE)
实际操作
1.创建新变量
变量名 <- 表达式
mydata <- transform(mydata,sumx = x1+x2,
meanx = (x1+x2)/2)
2.变量重编码
重编码:根据同一个变量和/或其他变量的现有值创建新值的过程。过程涉及逻辑判断。
- 连续变量修改为类别值
- 误编码的值修改为正确值
- 根据某一个标准创建新变量
leadership$age[leadership$age == 99] <- NA
leadership <- within(leadership,{
agecat <- NA
agecat[age >75] <- "Elder"
agecat[age >= 55 && age <=75 ] <- "Middle Aged"
agecat[age <55] <- "Young"
})
一些重编码函数:car包的recode(),doBy包的recodevar(),自带的cut()
3.变量重命名
names(leadership)[2] <- "testDate
推荐:dplyr::rename
4.缺失值
缺失值以NA表示。
判断:is.na
注意:缺失值不可比较,无法用比较运算符检测;R语言有专门符号来表示无限大(+ inf,-inf)和不可能的值(NaN)
分析中注意各个函数对缺失值的处理,可以使用na.omit()删除所有含有缺失数据的行。
5.日期值
使用as.Date()将字符型日期值变为数值形式
使用as.character()将数值形式的日期变成字符型
6.类型转换
R语言中提供了大量用于判断某个对象的数据类型和转换方式
| 判断 | 转换 |
|---|---|
| is.numeric() | as.numeric() |
| is.character() | as.character() |
| is.vector() | as.vector() |
| is.matrix() | as.matrix() |
| is.data.frame() | as.data.frame() |
| is.factor() | as.factor() |
| is.logical() | as.logical() |
7.数据排序
根据年龄进行排序
newdata <- leadership[oerder(leadership$age)]
8.数据集合并
- 向数据框添加列
total <- merge(dateframeA,dataframeB,by="ID")
- 向数据框添加行
一定要有相同的变量
total <- rbind(dataframeA,dataframeB)
9.数据集取子集
- 选入(保留)变量
#data.frame[row indices, column indices]
newdata <- leadership[,c(6:10)]
- 剔除(丢弃)变量
例如,剔除q3和q4列
# 法1
newvar <- names(leadership) %in% c("q3","q4")
newdata <- leadership[!newvar]
# 法2
newdata <- leadership[c(-8,-9)]
# 法3
leadership$q3 <- leadership$q4 <- NULL
- 选入观测
newdata <- leadership[1:3,]
newdata <- leadership[leadership$gender =="M" & leadership$age > 30,]
- subset函数
比较简单的方法:
newdata <- subset(leadership,age >= 35 | age <24, select=c(q1,q2,q3,q4))
- 随机抽样
通过抽样,用其中一份构建预测模型,使用一份验证模型的可靠性
mydata <- leadership[sample(1:nrow(leadership),3,replace=FALSE),]
高级数据管理
目标:
- 数学和统计函数
- 字符处理函数
- 循环和条件执行
- 自编函数
- 数据整合与重塑
基石函数
一些重要函数在《R语言实战》(第二版)的页码:
- 数学函数,P86-87
- 统计函数,P87-88
- 概率函数,P90
- 字符函数,P93
- 其他实用函数, P94
自编函数
除了这些基石函数外,R语言还支持大量的扩展包,以及用户自编函数用于处理某个特定的问题
myfunction <- function(arg1,arg2,...){
statements
return(object)
}
可以在函数中添加控制流,如重复和循环以及条件执行。
for (var in seq) statement
while (cond) statement
if (cond) statement
if (cond) statement1 else statement2
ifelse(cond, statement1 statement2)
这些由于不是本文的重点,不详细展开。
应用函数
如何将上述这些函数应用(apply)到矩阵、数组、数据框的任何维度上呢?
R语言提供了除了循环语句外更好的选择:apply,sapply,lapply
其中apply作用于相同数据类型的数据结构(如vector,matrix,array),而sapply和lapply作用与含有不同数据类型的数据结构(如data.frame,list)
1.apply的用法
apply的一般形式:
apply(m,dimcode,f,fargs)
# m是矩阵或数组或数据框
# dimcode, 维度编号,1代表行,2代表列
# f, 应用函数
# fargs f的可选参数
例如对每列求和
set.seed(941122)
mx <- matrix(runif(50),nrow=5)
apply(mx,2,sum)
注意到还有一个fargs,在调用的函数需要多于一个参数时使用,例如如果matrix中有NA,然而mean在默认下保留NA,有时候你需要主动声明。
mx[1,3] <- NA
apply(mx,2,mean)
apply(mx,2,mean,na.rm=TRUE)
如果数据库每一列的数据类型都相同,也就可以当做矩阵处理,以自带mtcar数据为例
apply(mtcars,2,mean)
2.lapply和sapply
lapply和sapply功能相同,区别就是lapply返回的依旧是list,而sapply返回的是vector,算是lapply的“友好版”
lst <- list(a=rnorm(6,mean=1),b=rnorm(6,mean=4),c=rnorm(6,mean=6))
lst.lapply <- lapply(lst,mean)
lst.sapply <- sapply(lst,mean)
3.mapply,vapply
还有两个不怎么常用的apply家族成员,mapply用于处理多层嵌套的list,vapply预先定义返回的内容,速度比较快,而且容易发现错误。自行查阅help().
整合和重构
整合和重构可能是一类比较高级的数据管理了。
整合:将多组观测替换为根据这些观测计算的描述性统计量
重塑:修改数据的结构(行和列)来决定数据的组织形式。
下面用mtcars来就具体说明。
热身:转置
转置可能是重塑数据集中最简单的啦,在Excel里面,你可以需要先选择一部分区域,然后复制的时候选择转置,R里面通过t()来实现
cars <- mtcars[1:5,1:4]
t(cars)
整合数据
在R中可以使用一个或多个by变量和一个预先定义好的函数来折叠(collapse)数据。调用格式为:
aggregate(x,by,FUN)
实例:
options(digits=3)
attach(mtcars)
aggrata <- aggragate(mtcars,by=list(cyl,gear),FUN=mean,na.rm=TRUE)
Group.1 Group.2 mpg cyl disp hp drat wt qsec vs am gear carb
1 4 3 21.5 4 120 97 3.70 2.46 20.0 1.0 0.00 3 1.00
2 6 3 19.8 6 242 108 2.92 3.34 19.8 1.0 0.00 3 1.00
3 8 3 15.1 8 358 194 3.12 4.10 17.1 0.0 0.00 3 3.08
4 4 4 26.9 4 103 76 4.11 2.38 19.6 1.0 0.75 4 1.50
5 6 4 19.8 6 164 116 3.91 3.09 17.7 0.5 0.50 4 4.00
6 4 5 28.2 4 108 102 4.10 1.83 16.8 0.5 1.00 5 2.00
7 6 5 19.7 6 145 175 3.62 2.77 15.5 0.0 1.00 5 6.00
8 8 5 15.4 8 326 300 3.88 3.37 14.6 0.0 1.00 5 6.00
结果根据气缸数目(group.1)和档位数(group2)对求不同分组的平均值
强大无比的tidyr包
reshpe2包是一套重构和整合数据集的绝妙的万能工具,而tidyr是他的继承者。
加载到R语言中的表格数据有两种类型,long和wide.例如:
- wide format
| 基因 | 分生组织 | 根 | 花 |
|---|---|---|---|
| gene1 | 582 | 91 | 495 |
| gene2 | 305 | 3505 | 33 |
- long format
| 基因 | 组织 | 表达量 |
|---|---|---|
| gene1 | 分生组织 | 582 |
| gene2 | 分生组织 | 305 |
| gene1 | 根 | 91 |
| gene2 | 根 | 3503 |
| gene1 | 花 | 492 |
| gene2 | 花 | 33 |
一般而言我们记录数据的格式通常是long format,但是实际上机器比较喜欢的wide format,为了让机器能够更好理解数据,我们需要在内部把long format 转成 wide format. 这里用的就是tidyr。
这么牛逼好用的包是谁开发的呢,是Hadley Wickham!.
 Hadley Wickham
Hadley Wickham
tidyr使用gather()和spread()来改变数据储存的格式,类似于前任reshape2的melt()和cast(),如下图所示:
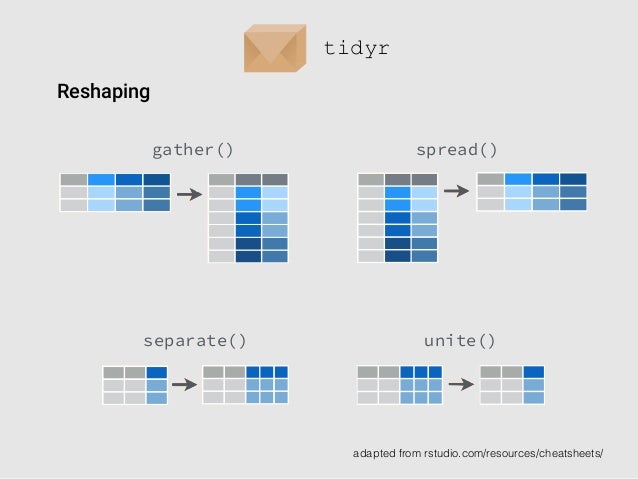
具体操作
- 将wide format,转成long format
数据:
gene <- c("gene1","gene2")
meristem <- c(582,305)
root <- c(91,3505)
flower <- c(495,33)
gene.expr <- data.frame(gene=gene,meristem=meristem,root=root,flower=flower)
用法:
gather(data, key, value, ..., na.rm = FALSE, convert = FALSE,factor_key = FALSE)
data:用于转换的数据框,key和value分别对应转换后结果,...就是用于进行转换的列名(-A表示剔除列A,A:B,表示从A到B)。
long.format <-gather(data = gene.expr,key = Tissue, value = Expression,meristem:flower )
gene Tissue Expression
1 gene1 meristem 582
2 gene2 meristem 305
3 gene1 root 91
4 gene2 root 3505
5 gene1 flower 495
6 gene2 flower 33
- 将long format转成wide format
当数据处理结束后,数据的结果毕竟是给人看,可以用spread()进行转换。
spread(data, key, value, fill = NA, convert = FALSE, drop = TRUE,sep = NULL)
含义同上。
spread(long.format,key=Tissue,Value=Expression)
gene flower meristem root
1 gene1 495 582 91
2 gene2 33 305 3505
结语
以上是R语言中数据操作基本知识,下一部分介绍如何利用这些基本知识,进行数据探索性分析(Exploratory Data Analysis, EDA)
参考资料:
- [R语言实践]
- [Bioinformatics Data Skills]















网友评论