用CNN识别CT图像检测肺癌
原文:2nd place solution for the 2017 national datascience bowl
翻译参考:知乎用户王小新 Kaggle百万美元大赛优胜者:用CNN识别CT图像检测肺癌
概要
本文为2017年由Kaggle举办的数据科学竞赛的第二名获奖者Julian de Wit的部分解决方案。Julian de Wit和Daniel Hammack通过构建3D卷积神经网络创建肺结节探测器,预测患者患癌的可能性。Wit的开发环境为Windows版的Tensorflow和Keras库。
Kaggle在该比赛中提示参赛选手可参考之前的LUNA16竞赛。该竞赛采用的数据集是由公开数据集LIDC-IDRI转化而来的,医生为800多个病人的CT图像标记了1000多个肺结节。因此,可以从整张CT图像中的标记周围裁剪出小型3D图像,将这些小型3D图像与结节标记直接对应,从而利用神经网络学习这些特征,训练出一个神经网络来检测肺结节,并评估结节的恶性程度,预测患者患癌的可能性。其中,预测时神经网络通过滑动窗口的方式来遍历整张CT图像,分别判断每个滑动窗口所包含的区域是否含有恶性信息的可能性。
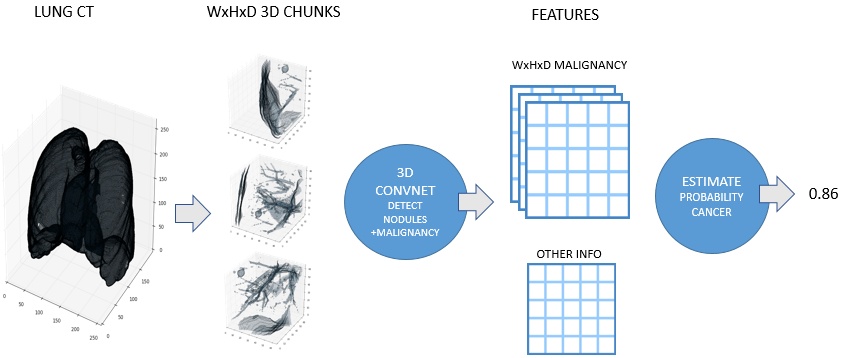
数据预处理和创建训练集
数据预处理
在预处理过程中,首先对CT图像进行缩放,保证图像中的每个像素点只表示1mm3的体积;然后将CT图像的像素强度转换为HU值,并最大化HU值后进行归一化处理;最后,确保所有CT图像都具有相同的方向。
训练集构建
计划构建U-net网络分割肺部区域。根据观察CT图像,可利用肺组织的边缘构建相应的框架找到肺结节。
 带有标记的不同数据集
带有标记的不同数据集
在分割掩膜边缘进行采样标注,从而分割得到肺部组织。
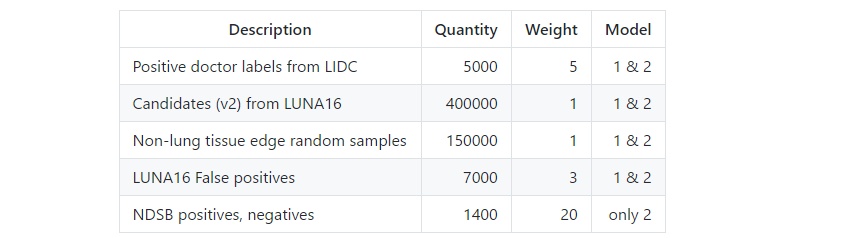
在第一轮训练中,对LUNA16数据集上进行结节预测,得到了所有假阳性结节,并将其并入训练集中。随后,手动标记NDSB数据集中癌症样本的阳性结节和非癌症样本的假阳性结节,并训练第二各模型,其效果很糟糕。但将两个模型相结合,其效果相比于单个模型较好,因此保留第二个模型。
建立一个结节观测器,用于调试所有的标记。LIDC数据集的说明文档表明医生被要求忽略大于3cm的结节,但担心这些被忽略的结节会影响分类器的准确度,从而删除了与这些结节相重叠的部分。
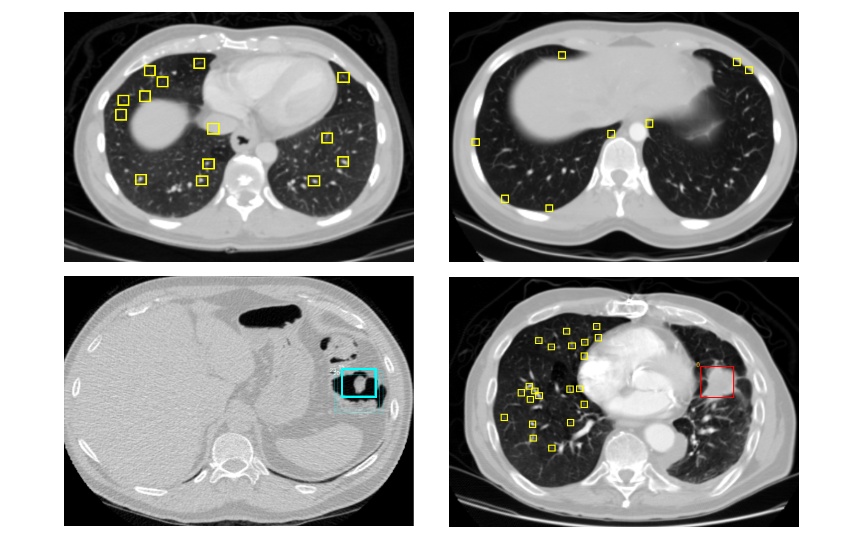 CT图像中的标记
CT图像中的标记
左上为LUNA16 V2的数据;右上为非肺组织的边缘;左下为假阳性的区域;右下为被移除的无标注区域。
3D卷积神经网络的训练方法和网络结构
数据集正反两类样本量比为5000:500000,且正面例子的大小和形状有很大差异。因此在CT图像的滑动窗口中,建立小型3D卷积神经网络。
第一个目标为训练一个可作为基础的结节检测器。首先对正例进行升采样(upsample),将正反两类的样本比上调至1:20;然后进行一些图像增强操作以提高模型的泛化能力。
设计好分类器后,再训练一个用于预测恶化程度的回归模型,将肿瘤恶化程度划分为1(很可能不是恶性)~5(很可能是恶性),且为了强调肿瘤的恶化程度,对之前的划分平方运算后将范围扩大到0~25。计划使用同一个网络,以多任务学习的方法,同时进行分类结点和估计恶化程度这两个任务。
采用基于C3D神经网络得到最终的分类评估网络。由于Daniel的网络输入为64×64×64mm,首先将输入大小设置为32×32×32mm,使网络的输出互补;对Z轴进行average pooling操作,使得每个体素表示2mm3的区域;最后,在网络的终端引入64个节点的全连接层。
注:此处不是直接预测恶性肿瘤,而是输出结节的恶化程度。
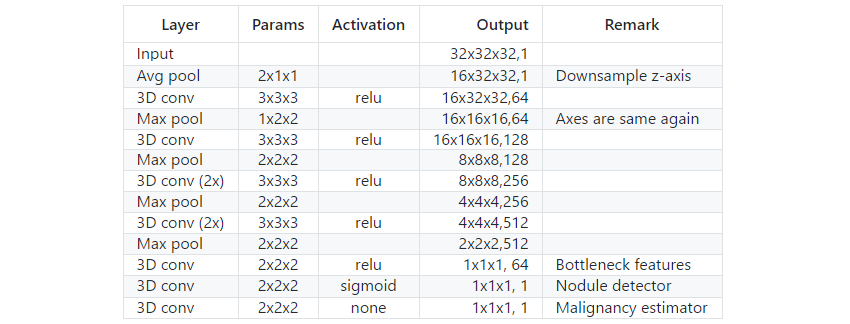 3D卷积神经网络结构示意图
3D卷积神经网络结构示意图
奇怪组织(strange tissue)检测器
当肺结节癌变时,其可能变为一些肺部肿块或更为复杂的组织。在CT图像中,若存在大量的“奇怪组织”,则表明癌症的可能性较高。然而,在目前的结节检测器程序中,对存在大量“奇怪组织”的CT图像,其无法检测出任意结节。因此,该问题将造成较高的假阳性率。
对于该问题,在LUNA16训练集上采用U-net网络构建检测器,但其效果提升微小。
癌症预测
在之前的处理过程中,存在一个严重问题:错过了一些较大的明显的结节。因此,对CT图像进行两次下采样(downsample),并让网络在1,1.5和2这三个尺度上预测。
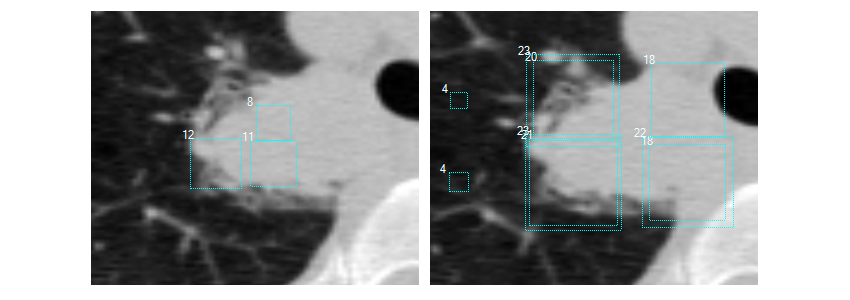
上图中左图表明没有很好地检测到大结节,图像缩放为1倍大小;右图表明检测效果较好,图像放大为2倍大小。图中矩形框表示检测到的恶性肿瘤。
添加额外的特征,构建梯度增强分类器(共使用7个特征)来预测一年内患者是否患癌。
主要由两个模型组成:
- 第一个模型基于所有的LUNA16数据集构建而成;
- 第二个模型通过选择NDSB3数据集中疑难病例和假阳性病例主动学习构建而成。





网友评论