在群体遗传学研究中,LD连锁不平衡分析是最常见的分析内容,也是关联分析的基础。如何正确理解并且进行相关的LD连锁不平衡分析是群体遗传中很基本的一件事情。下面和大家一起学习一下其相关的知识。
基础概念
如果要理解LD衰减图,我们就必须先理解连锁不平衡(Linkagedisequilibrium,LD)的概念。连锁不平衡是由两个名词构成,连锁+不平衡。前者,很容易让我们产生概念混淆;后者,让这个概念变得愈加晦涩。因此从一个类似的概念入手,大家可能更容易理解LD的概念,那就是基因的共表达。换句话来说,当位于某一座位的特定等位基因与另一座位的某一等位基因同时出现的概率大于群体中因随机分布的两个等位基因同时出现的概率时,就称这两个座位处于连锁不平衡状态(linkage disequilibrium)。
如果两个SNP标记位置相邻,那么在群体中也会呈现基因型步调一致的情况。比如有两个基因座,分别对应A/a和B/b两种等位基因。如果两个基因座是相关的,我们将会看到某些基因型往往共同遗传,即某些单倍型的频率会高于期望值。
例如在下图2中,在群体中(A,a,B,b)各个基因型的频率已知的情况下,各种单倍型的期望频率(AB、Ab、aB、ab)都是可以计算出来。例如,AB的频率=(A的频率)X(B的频率)。但我们实际统计群体中各个单倍型的频率的时候,会观察到某些单倍型的频率会大于期望值,例如下图中的单倍型AB的理论频率是0.12,但观察到的实际频率是0.29。那么说明,基因型A更倾向于基因型B共同遗传。

这种不同基因座间的相关性,用一个数值来衡量就是D值(图2中有计算公式)。类似相关系数是标准化后的协方差,LD系数(r2)则是标准化后的D值(图2中有计算公式),这个数值在0~1波动。r2=0就是两个位点完全不相关,群体中单倍型分布是随机的(观测值=期望值)。r^2=1就是两个位点完全相关,某些基因型(A)只与特定的基因型(B)共同出现。
一般而言,两个位点在基因组上离得越近,相关性就越强,LD系数就越大。反之,LD系数越小。也就是说,随着位点间的距离不断增加,LD系数通常情况下会慢慢下降。这个规律,通常就会使用LD衰减图来呈现。
图形理解和应用

LD衰减图就是利用曲线图来呈现基因组上分子标记间的平均LD系数随着标记间距离增加而降低的过程。大概的计算原理就是先统计基因组上两两标记间的LD系数大小,再按照标记间的距离对LD系数进行分类,最终可以计算出一定距离的分子标记间的平均LD系数大小。如图3是黄瓜重测序文章中统计各个亚群体的LD衰减速度的图形。横坐标是物理距离(kb),纵坐标是LD系数(r^2)。
从图中我们可以看出,西双版纳这个亚群体(紫色线)在基因组上50kb距离的平均LD系数大小约为0.4,但到了100kb的距离,对应的平均LD系数大小则降低到了不到0.3。而且,我们从图中也可以观察到LD系数的衰减速度在不同的亚群体快慢不同,衰减速度是 india > East Asian& Eurasian > Xishuanbanna。那说明india群体的LD衰减距离最小,可能是india这个群体遗传多样性最高导致。
LD衰减距离
实际上,LD衰减的速度在不同物种间或同物种的不同亚群体间,往往差异非常巨大。所以,通常会使用1个标准——“LD衰减距离”来描述LD衰减速度的快慢。
LD衰减距离通常指的是:当平均LD系数衰减到一定大小的时候,对应的物理距离。
“一定大小”是这个定义的关键点,但没有特别统一的标准,在不同文章中标准不同。常见的标准包括:a)LD系数降低到最大值的一半;b)LD系数降低到0.5以下;c)LD系数降低到0.1以下;d)LD系数降低到基线水平(但注意,不同材料的基线值是不同的。比如图3黄瓜群体的基线大概是0.1)。
LD衰减影响因素
LD系数衰退速度会受到不同因素的影响而有所不同。常见的因素包括:
1)物种类型LD存在的本质是两个位点的连锁遗传导致的相关性。但这种相关性理论上会随着世代的增加、重组次数的增加而不断下降。所以,那些繁殖力强、时代间隔短的物种(例如,昆虫),其LD衰减的速度是非常快的。例如在家蚕和野蚕群体中,LD系数下降到最大值的1/2仅仅需要46bp和7bp的距离
2)群体类型相同物种的不同群体,由于其遗传背景不同,LD衰减速度也存在很大的差异。驯化选择,会导致群体遗传多样性下降,位点间的相关性(连锁程度)加强。所以,通常驯化程度越高,选择强度越大的群体,LD衰减速度是最慢的。例如,栽培稻比野生稻通常更大的LD衰减距离。类似的,自然选择、遗传漂变导致的群体遗传多样性下降,也会减慢LD衰减的速度。
3)在染色体的位置染色体不同区域的LD衰减距离而是不同的。通常着丝粒区更难重组,所以LD衰减更慢。而基因组上那些受选择的区域相比普通的区域,LD衰减速度也是更慢的。
一般而言,LD系数大于0.8就是强相关。如果LD系数小于0.1,则可以认为没有相关性。如果LD衰减到0.1这么大的区间内都没有标记覆盖的话,即使这个区间有一个效应很强的功能突变,也是检测不到关联信号的。所以,通常可以通过比较LD衰减(到0.1)距离和标记间的平均距离,来判断标记是否对全基因组有足够的覆盖度。(GWAS标记量=基因组大小/LD衰减距离)
实战分析
这里会用到华大研发的一款软件PopLDdecay。
下载安装
git clone https://github.com/BGI-shenzhen/PopLDdecay.git
chmod 755 configure;
./configure;
make;
mv PopLDdecay bin/;
基本使用说明:
Usage: PopLDDecay -InVCF <in.vcf.gz> -OutStat <out.stat>
-InVCF <str> Input SNP VCF Format
-InGenotype <str> Input SNP Genotype Format
-OutStat <str> OutPut Stat Dist ~ r^2 File
-SubPop <str> SubGroup SampleList of VCFFile [ALLsample]
-MaxDist <int> Max Distance (kb) between two SNP [300]
-MAF <float> Min minor allele frequency filter [0.005]
-Het <float> Max ratio of het allele filter [0.88]
-Miss <float> Max ratio of miss allele filter [0.25]
-EHH <str> To Run EHH Region decay set StartSite [NA]
-OutFilterSNP OutPut the final SNP to calculate
-OutType <int> 1: R^2 result 2: R^2 & D'
这个工具可以对整个群体进行LD衰减图绘制:
./PopLDdecay -InVCF overlap.filter.vcf -OutStat overlap.all.stat
#运行绘图需要你系统内安装好R
perl Plot_OnePop.pl -inFile overlap.all.stat -output all.grpah
来看看效果如何,基本是一条圆滑的曲线,趋势也是比较符合:
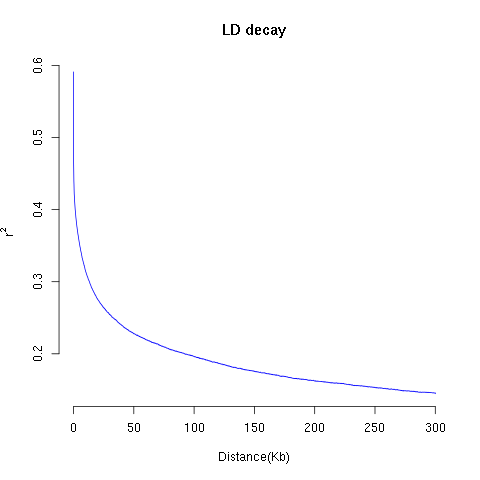
接着安装不同的群体来进行LD衰减图的绘制:
#分别对不同的群体进行LD分析:
./PopLDdecay -InVCF overlap.filter.vcf -SubPop lan.txt -OutStat overlap.lan.stat
./PopLDdecay -InVCF overlap.filter.vcf -SubPop cul.txt -OutStat overlap.cul.stat
./PopLDdecay -InVCF overlap.filter.vcf -SubPop wild.txt -OutStat overlap.wild.stat
./PopLDdecay -InVCF overlap.filter.vcf -SubPop adm.txt -OutStat overlap.adm.stat
#进行图形的绘制:
perl Plot_MultiPop.pl -inList draw.list -output draw.graph
###这里对于这个-inList的输入格式需要注意一下(stat的path然后加上你stat文件的前缀),可以参考我的输入文件:
/scratch/pawsey0149/hhu/tool/PopLDdecay/bin/overlap.adm.stat.gz overlap.adm
/scratch/pawsey0149/hhu/tool/PopLDdecay/bin/overlap.cul.stat.gz overlap.cul
/scratch/pawsey0149/hhu/tool/PopLDdecay/bin/overlap.lan.stat.gz overlap.lan
/scratch/pawsey0149/hhu/tool/PopLDdecay/bin/overlap.wild.stat.gz overlap.wild
好继续看看结果如何,符合上面说到的,野生种具有最慢的衰减速度,因为其多样性最多,接着是地方种,然后到杂交种,最后是栽培种:
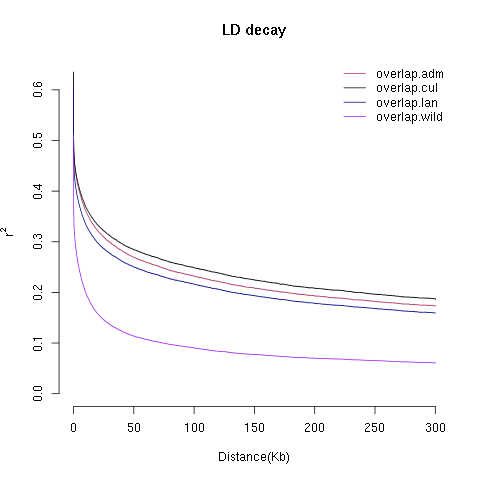 draw.graph.png-17.6kB
draw.graph.png-17.6kB
基础部分是摘抄于基迪奥的论坛,因为本来它那里已经说得很清楚明白了,直接引用就好了,我觉得没必要再造车,下次会继续探讨一下LD block的相关知识。
参考资料:















网友评论