tags: Trimmomatic NGS fastq
NGS 原始数据过滤对后续分析至关重要,去除一些无用的序列也可以提高后续分析的准确率和效率。Trimmomatic 是一个功能强大的数据过滤软件。
Trimmomatic 介绍
Trimmomatic 发表的文章至今已被引用了 2810 次,是一个广受欢迎的 Illumina 平台数据过滤工具。其他平台的数据例如 Iron torrent ,PGM 测序数据可以用 fastx_toolkit 、NGSQC toolkit 来过滤。
Trimmomatic 支持多线程,处理数据速度快,主要用来去除 Illumina 平台的 Fastq 序列中的接头,并根据碱基质量值对 Fastq 进行修剪。软件有两种过滤模式,分别对应 SE 和 PE 测序数据,同时支持 gzip 和 bzip2 压缩文件。
另外也支持 phred-33 和 phred-64 格式互相转化,现在之所以会出现 phred-33 和 phred-64 格式的困惑,都是 Illumina 公司的锅(damn you, Illumina!),不过现在绝大部分 Illumina 平台的产出数据也都转为使用 phred-33 格式了。
Trimmomatic 过滤步骤
Trimmomatic 过滤数据的步骤与命令行中过滤参数的顺序有关,通常的过滤步骤如下:
- ILLUMINACLIP: 过滤 reads 中的 Illumina 测序接头和引物序列,并决定是否去除反向互补的 R1/R2 中的 R2。
- SLIDINGWINDOW: 从 reads 的 5' 端开始,进行滑窗质量过滤,切掉碱基质量平均值低于阈值的滑窗。
- MAXINFO: 一个自动调整的过滤选项,在保证 reads 长度的情况下尽量降低测序错误率,最大化 reads 的使用价值。
- LEADING: 从 reads 的开头切除质量值低于阈值的碱基。
- TRAILING: 从 reads 的末尾开始切除质量值低于阈值的碱基。
- CROP: 从 reads 的末尾切掉部分碱基使得 reads 达到指定长度。
- HEADCROP: 从 reads 的开头切掉指定数量的碱基。
- MINLEN: 如果经过剪切后 reads 的长度低于阈值则丢弃这条 reads。
- AVGQUAL: 如果 reads 的平均碱基质量值低于阈值则丢弃这条 reads。
- TOPHRED33: 将 reads 的碱基质量值体系转为 phred-33。
- TOPHRED64: 将 reads 的碱基质量值体系转为 phred-64。
Trimmomatic 简单用法
由于 Trimmomatic 过滤数据的步骤与命令行中过滤参数的顺序有关,因此,如果需要去接头,建议第一步就去接头,否则接头序列被其他的过滤参数剪切掉部分之后就更难匹配更难去除干净了。
单末端测序模式
在 SE 模式下,只有一个输入文件和一个过滤之后的输出文件:
java -jar <path to trimmomatic jar> SE [-threads <threads>] [-phred33 | -phred64] [-trimlog
<logFile>] <input> <output> <step 1> <step 2> ...
-trimlog 参数指定了过滤日志文件名,日志中包含以下四列内容:
- read ID
- 过滤之后剩余序列长度
- 过滤之后的序列起始碱基位置(序列开头处被切掉了多少个碱基)
- 过滤之后的序列末端碱基位置
- 序列末端处被剪切掉的碱基数
由于生成的 trimlog 文件中包含了每一条 reads 的处理记录,因此文件体积巨大(GB 级别),如果后面不会用到 trim 日志,建议不要使用这个参数。
双末端测序模式
在 PE 模式下,有两个输入文件,正向测序序列和反向测序序列,但是过滤之后输出文件有四个,过滤之后双端序列都保留的就是 paired,反之如果其中一端序列过滤之后被丢弃了另一端序列保留下来了就是 unpaired 。
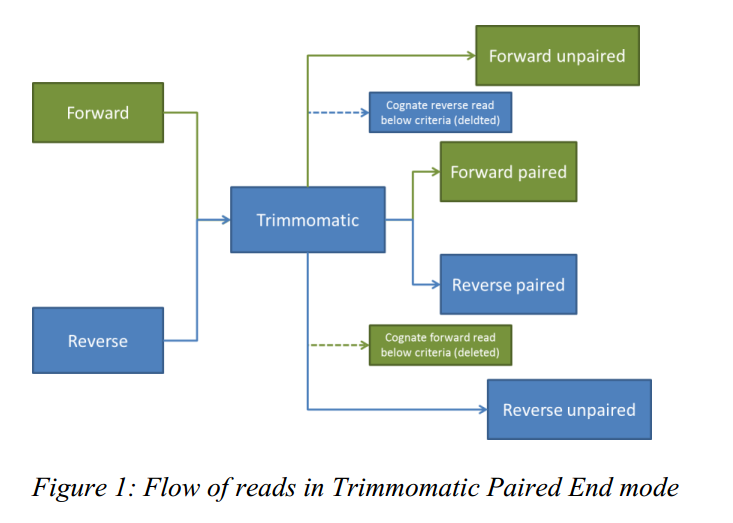 PE 模式下的输入输出文件
PE 模式下的输入输出文件
java -jar <path to trimmomatic.jar> PE [-threads <threads] [-phred33 | -phred64] [-trimlog
<logFile>] >] [-basein <inputBase> | <input 1> <input 2>] [-baseout <outputBase> |
<paired output 1> <unpaired output 1> <paired output 2> <unpaired output 2> <step 1> <step 2> ...
其中 -phred33 和 -phred64 参数指定 fastq 的质量值编码格式,如果不设置这个参数,软件会自动判断输入文件是哪种格式(v0.32 之后的版本都支持),虽然软件默认的参数是 phred64,如果不确定序列是哪种质量编码格式,可以不设置这个参数。
输入输出文件
PE 模式的两个输入文件:sample_R1.fastq sample_R2.fastq以及四个输出文件:sample_paired_R1.clean.fastq sample_unpaired_R1.clean.fastq sample_paired_R1.clean.fastq sample_unpaired_R1.clean.fastq
通常 PE 测序的两个文件,R1 和 R2 的文件名是类似的,因此可以使用 -basein 参数指定其中 R1 文件名即可,软件会推测出 R2 的文件名,但是这个功能实测并不好用,因为软件只能自动识别推测三种种格式的 -basein:
- Sample_Name_R1_001.fq.gz -> Sample_Name_R2_001.fq.gz
- Sample_Name.f.fastq -> Sample_Name.r.fastq
- Sample_Name.1.sequence.txt -> Sample_Name.2.sequence.txt
建议不用 -basein 参数,直接指定两个文件名(R1 和 R2)作为输入。
输出文件有四个,当然也可以像上文一样指定四个文件名,但是参数太长有点麻烦,有个省心的方法,使用 -baseout 参数指定输出文件的 basename,软件会自动为四个输出文件命名。例如 -baseout mySampleFiltered.fq.gz ,文件名中添加 .gz 后缀,软件会自动将输出结果进行 gzip 压缩。输出的四个文件分别会自动命名为:
- mySampleFiltered_1P.fq.gz - for paired forward reads
- mySampleFiltered_1U.fq.gz - for unpaired forward reads
- mySampleFiltered_2P.fq.gz - for paired reverse reads
- mySampleFiltered_2U.fq.gz - for unpaired reverse reads
此外,如果直接指定输入输出文件名,文件名后添加 .gz 后缀就是告诉软件输入文件是 .gz 压缩文件,输出文件需要用 gzip 压缩。
过滤原理
每一步的过滤如果需要多个参数,通常用冒号:将各个参数隔开,当然参数的先后顺序是有要求的。
ILUMINACLIP
从名字可以看出,这一步是为了去除 illumina 接头的,这个软件其实就是专为 illumina 平台数据而设计的。
为了更好理解测序 reads 中为什么会有引物和接头序列,我画了一个文库加上接头之后的结构示意图,也把引物结合部位大概标了出来:
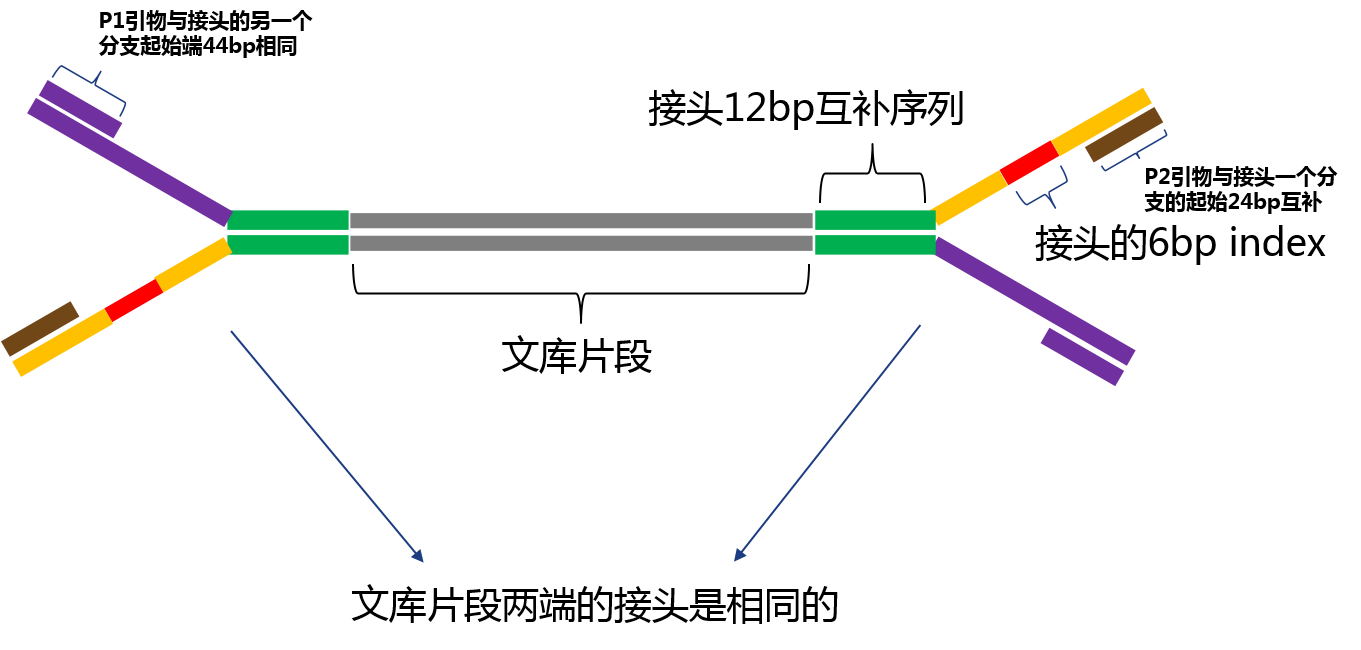 接头和测序引物结构示意图
接头和测序引物结构示意图
这个文库结构示意图理解之后就容易理解测序过程了。
去除接头以及引物序列看似简单,但需要权衡灵敏度(保证接头和引物去除干净)和特异性(保证不是接头和引物的序列不被误切除),由于测序中可能存在的随机错误让去接头这样一个简单的操作变的复杂。
虽然理论上接头序列和引物序列可能出现在 reads 中的任何位置,但实际上序列中出现接头和引物大部分情况下都是由于文库插入片段比测序读长短导致的,这种情况在 reads 的开头部分是有一段可用序列的,末端包含了接头的全长或部分序列,如果末端只有接头的一部分序列,那么去除这残缺的接头序列也不是容易的事。
然而,在 PE 测序模式下如果文库的插入片段比测序读长短,那么 read1 和 read2 中非接头序列的那部分会完全反向互补,Trimmomatic 有一个 ‘palindrome’ 模式会利用这个特点进行接头序列的去除。
下图中 A、B、C、D 四种情况就是 Trimmomatic 去除接头和引物的四种模式:
- 红色条形:被切除的序列
- 绿色条形:保留下来的有效读长
- 深蓝色条形:接头序列
- 浅蓝色条形:引物序列
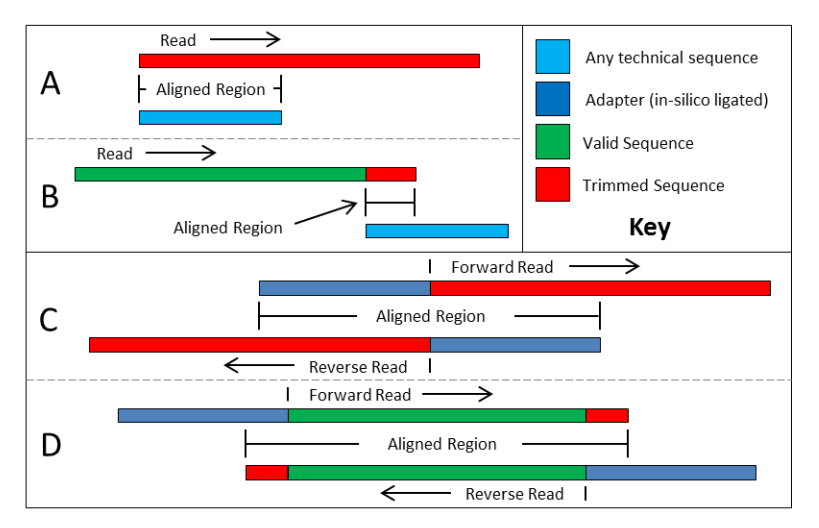 Trimmomatic 四种模式原理图解
Trimmomatic 四种模式原理图解
A 模式:测序 reads 从起始位置开始就包含了完整的接头序列,那么根据 Illumina 测序原理,这整条 reads 都不可能包含有用序列了,整条 reads 被丢弃。
B 模式:这种相对常见,由于文库插入片段比测序读长短,会在 reads 末端包含部分接头序列,若是这部分接头序列足够长是可以识别并去除的,但如果接头序列太短,比接头匹配参数设置的最短长度还短,那么就无法去除。但是,如果是 PE 测序,可以按照 D 模式去除 reads 末端的很短的接头序列。
C 模式:PE 测序可能出现这种情况,正向测序和反向测序有部分完全反向互补,但是空载的文库,两个接头直接互连,这样的 reads 不包含任何有用序列,正反向测序 reads 都被丢弃。
D 模式:是 Trimmomatic 利用 PE 测序进行短接头序列去除的典范,如果文库插入片段比测序读长短,利用正反向测序 reads 中一段碱基可以完全反向互补的特点,将两个接头序列与 reads 进行比对,同时两条 reads 之间也互相比对,可以将 3' 末端哪怕只有 1bp 的接头序列都可以被准确去除,相对 B 模式去除接头污染更彻底。
Trimmomatic 使用了一种类似序列比对软件(例如 Isaac aligner,一个超快速的 alignment 软件)的两步策略来搜索潜在的接头序列。首先,使用接头序列中的一段种子序列(seed 长度不超过 16bp)与测序 reads 进行比对,如果种子序列在测序 reads 中有足够好的比对结果(具体由 seedMismatch 参数决定),就启动第二步的接头全长与 reads 比对。第一步的 seed 搜索速度很快,可以过滤掉没有接头污染的 reads ,这种两步搜索的方法使得接头序列的查找效率很高。
在第二步的接头序列和测序 reads 全长比对统计比对分值时,罚分策略考虑了测序碱基的质量值Q,每一个比对上的碱基加分 0.6,每一个错配的碱基减分 Q/10,考虑碱基质量值可以降低低质量碱基(高测序错误率)错配对整个比对得分的影响。在这个规则下,一段 12bp 的接头序列完全比对到 reads 上得分为 7.2, 25bp 的接头序列完全比对到 reads 上得分为 15。因此在 ILLUMINACLIP 参数中 simple clip threshold 的值建议为 7-15 之间(即上图中 A/B 比对模式比对得分阈值)。
对于 palindromic 模式的比对(上图中 D 模式),可以比对上的序列长度会更长,为了保证识别接头序列的准确率,比对得分的阈值也更高,例如 reads的 R1 和 R2 中有 50bp 序列可以反向互补匹配,得分为 30。这种模式下,Trimmomatic 可以识别并去除 reads 中非常短的接头序列。
ILLUMINACLIP 参数说明:按照规定顺序,ILLUMINACLIP 的参数列表如下(各个参数之间以冒号分开),PE 测序需要注意最后一个参数。对于 SE 测序最后两个参数可以不设置。
ILLUMINACLIP:<fastaWithAdaptersEtc>:<seed mismatches>:<palindrome clip threshold>:<simple clip threshold>:<minAdapterLength>:<keepBothReads>
ILLUMINACLIP:TruSeq3-SE:2:30:10 #接头和引物序列在 TruSeq3-SE 中,第一步 seed 搜索允许2个碱基错配,palindrome 比对分值阈值 30,simple clip 比对分值阈值 10,palindrome 模式允许切除的最短接头序列为 8bp(默认值),palindrome 模式去除与 R1 完全反向互补的 R2(默认去除)
fastaWithAdaptersEtc:指定包含接头和引物序列(所有被视为污染的序列)的 fasta 文件路径,Trimmomatic 自带了一个包含 Illumina 平台接头和引物序列的 fasta 文件,可以直接用这个。
seedMismatches:指定第一步 seed 搜索时允许的错配碱基个数,例如 2。
palindrome clip threshold:指定针对 PE 的 palindrome clip 模式下,需要 R1 和 R2 之间至少多少比对分值(上图中 D 模式),才会进行接头切除,例如 30。
simple clip threshold:指定切除接头序列的最低比对分值(上图 A/B 模式),通常 7-15 之间。
minAdapterLength:只对 PE 测序的 palindrome clip 模式有效,指定 palindrome 模式下可以切除的接头序列最短长度,由于历史的原因,默认值是 8,但实际上 palindrome 模式可以切除短至 1bp 的接头污染,所以可以设置为 1 。
keepBothReads:只对 PE 测序的 palindrome clip 模式有效,这个参数很重要,在上图中 D 模式下, R1 和 R2 在去除了接头序列之后剩余的部分是完全反向互补的,默认参数 false,意味着整条去除与 R1 完全反向互补的 R2,当做重复去除掉,但在有些情况下,例如需要用到 paired reads 的 bowtie2 流程,就要将这个参数改为 true,否则会损失一部分 paired reads。
看一个 PE150 数据的测试,就知道 keepBothReads 参数的重要性了:
$ java -jar trimmomatic-0.36.jar PE -phred33 F-2-test_R1.fastq.gz F-2-test_R2.fastq.gz -baseout F-2.fastq.gz ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:51
ILLUMINACLIP: Using 1 prefix pairs, 0 forward/reverse sequences, 0 forward only sequences, 0 reverse only sequences
Input Read Pairs: 2500 Both Surviving: 1633 (65.32%) Forward Only Surviving: 828 (33.12%) Reverse Only Surviving: 12 (0.48%) Dropped: 27 (1.08%)
TrimmomaticPE: Completed successfully
# 使用 ILLUMINACLIP 默认的第六个参数 false,只有 65.32% paired reads 保留下来
$ java -jar trimmomatic-0.36.jar PE -phred33 F-2-test_R1.fastq.gz F-2-test_R2.fastq.gz -baseout F-2.fastq.gz ILLUMINACLIP:TruSeq3-PE.fa:2:30:10:8:true LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:51
ILLUMINACLIP: Using 1 prefix pairs, 0 forward/reverse sequences, 0 forward only sequences, 0 reverse only sequences
Input Read Pairs: 2500 Both Surviving: 2439 (97.56%) Forward Only Surviving: 22 (0.88%) Reverse Only Surviving: 16 (0.64%) Dropped: 23 (0.92%)
TrimmomaticPE: Completed successfully
# 将 ILLUMINACLIP 第六个参数改为 true,其余所有参数均相同,结果有 97.56% paired reads 保留下来
SLIDINGWINDOW
滑窗剪切,统计滑窗口中所有碱基的平均质量值,如果低于设定的阈值,则切掉窗口。
SLIDINGWINDOW 参数如下:
SLIDINGWINDOW:<windowSize>:<requiredQuality>
SLIDINGWINDOW:4:15 #设置 4bp 窗口,碱基平均质量值阈值 15
widowSize:设置窗口大小
requiredQuality:设置窗口碱基平均质量阈值
MAXINFO
包含一个可以自动调整的过滤条件,在保留尽可能长的序列和保持序列中碱基测序错误率尽可能低之间进行平衡,以达到 trim 之后保留序列的价值最大化。
对于不同的应用场景,一条 reads 序列的价值由以下三个因素决定:
- 允许的最短 read 长度:只有在测序 reads 足够长的情况下,才能将 reads map 到参考序列的唯一位置上。过短的序列往往能 map 到参考基因组上的多个位置(特异性差),这种非特异性的 map 结果可以给出的有价值信息很少(对后续分析没什么用)。保证 reads 可以 map 到参考序列的唯一位置上需要的 reads 最短长度也与参考序列本身的长度和序列复杂度有关,但是通常最短 reads 要求不低于 40bp。
- 需求的额外 reads 长度:在保证 reads 可以 map 到参考序列上的唯一位置的情况下, reads 序列越长,可能对后续分析越有利。当然,这也需要看应用场景,对于 RNA-seq 来说只需要统计某个位置 map 上的 reads 数目,这种情况下 reads 长度只要满足唯一比对要求即可。而对于 de-novo 序列组装或者 Resequencing 检测变异,reads 序列长度越长越好,reads 越长可以为后续分析提供越多的有效证据。
- 对测序错误的敏感性:即测序错误率对后续分析工作是否造成严重影响,有的应用中对 reads 测序错误零容忍,就需要非常严格的过滤条件,而有的应用对测序错误不敏感,就可以使用宽松的过滤条件。
MAXINFO 有两个参数,第一个 target read length 控制上面的因素一,即允许的最短 read 长度。第二个参数 strictness 是控制因素二和因素三之间的平衡,即满足最短 read 长度的情况下,是保留尽可能多的碱基,还是保证尽可能低的测序错误率。
MAXINFO 过滤从 reads 3' 端开始进行剪切,在考虑上述三个因素的情况下统计所有可能的 trim 方式的到的 clean reads 的 INFO 分值(即所谓的 reads 价值),这三个因素分别以不同的方式影响最终的 reads INFO 分值:
- 最短 read 长度:最终保留的 reads 长度与 INFO 分值之间是 logistic 函数关系,即 INFO 分值与 reads 长度之间是 S 形增长曲线(类似大肠杆菌生长曲线),大概相当于,当保留下来的 reads 长度小于最短长度时,INFO 分值随着 reads 长度增加呈指数级增长,当保留下来的 reads 长度达到最短 read 长度要求之后,INFO 分值的增长会变缓。
- 超出最短 read 长度的部分:INFO 分值会随着额外的 reads 长度增加了线性增长,线性系数与 MAXINFO 的第二个参数有关,为 1 - strictness。
- 测序错误敏感性:最终保留下来的所有碱基的质量值都被用来计算 reads 包含测序错误的概率,出现测序错误的概率提升会降低 INFO 分值,错误概率对 INFO 分值的影响权重与 strictness 相关。
针对一条 read 的任何可能的剪切方式都计算出 INFO 分值,最终的 reads 长度和切除的碱基由 INFO 最大值决定。实际上这三个影响因子作用的方式不同:
- 当 reads 长度比最短 read 长度还短时 INFO 分值被序列长度主导,因为太短的 reads 根本无法提供足够的有用信息。
- 当 reads 长度满足最短 read 长度要求时,reads 的长度因素就不再是影响 INFO 分值的主导因素了,而且一旦 reads 长度足够长时,由于 logistic 函数的关系,reads 长度就不怎么贡献 INFO 分值了。
- 超出最短 read 长度要求的碱基对 INFO 贡献有限,而另一方面,由于碱基的增加导致 reads 中出现测序错误的概率增大,这会导致对 INFO 的罚分。控制这超出最短 read 长度的序列对 INFO 分值的影响到底是罚分还是得分,就看 strictness 参数了。
- 对于大部分足够长的 reads,保留超出最低要求的碱基序列是利还是弊,会依照碱基质量值分布和 strictness 参数来决定如何 trim。
参数说明:
MAXINFO:<targetLength>:<strictness>
targetLength:使得 reads 可以 map 到参考序列上唯一位置的最短长度(likely)。
strictness:一个介于 0 - 1 之间的小数,决定如何平衡 最大化 reads 长度 或者 最小化 reads 出现错误的概率,当参数设置小于 0.2 时倾向于最大化 reads 长度,当参数设置大于 0.8 时倾向于最小化 reads 中出现测序错误的概率。
LEADING
从 reads 的起始端开始切除质量值低于设定的阈值的碱基,直到有一个碱基其质量值达到阈值。
LEADING:<quality>
quality:设定碱基质量值阈值,低于这个阈值将被切除。
TRAILING
从 reads 的末端开始切除质量值低于设定阈值的碱基,直到有一个碱基质量值达到阈值。Illumina 平台有些低质量的碱基质量值被标记为 2 ,所以设置为 3 可以过滤掉这部分低质量碱基。官方推荐使用 Sliding Window 或 MaxInfo 来代替 LEADING 和 TAILING。
TRAILING:<quality>
quality:设定碱基质量值阈值,低于这个阈值将被切除。
CROP
不管碱基质量,从 reads 的起始开始保留设定长度的碱基,其余全部切除。一刀切,把所有 reads 切成相同的长度。
CROP:<length>
length:reads 从末端除之后保留下来的序列长度
HEADCROP
不管碱基质量,从 reads 的起始开始直接切除部分碱基。
HEADCROP:<length>
length:从 reads 的起始开始切除的碱基数
MINLEN
设定一个最短 read 长度,当 reads 经过前面的过滤之后,如果保留下来的长度低于这个阈值,整条 reads 被丢弃。被丢弃的 reads 数会被统计在 Trimmomatic 日志的 dropped reads 中。
MINLEN:<length>
length:可被保留的最短 read 长度
TOPHRED33
此选项可以将过滤之后的 Fastq 文件中质量值这一行转为 phred-33 格式。
TOPHRED64
此选项可以将过滤之后的 Fastq 文件中质量值这一行转为 phred-64 格式。
接头序列和引物序列
Trimmomatic 也可以自己制作包含接头和引物序列的 fasta 文件,格式可以参考软件自带的 adapters 文件夹中的格式。
adapters 文件夹中包含 illumina 测序 TruSeq2,TruSeq3 针对 SE 和 PE 的通用接头和引物序列。





网友评论