上传数据到 SRA 是个体力活,根据 Submission Portal 的指引一步一步填写相应的信息,需要上传的数据描述文件都要转为 TSV 格式再上传,测序数据先压缩再上传可以节省时间,不过只支持 gzip,bzip2 的压缩格式,不支持 .zip 和 .rar 压缩格式,可以使用 tar 进行打包。顺利的话传完数据填完信息提交就可以了,出现问题直接 Google 关键字。
填写项目信息表单的几个关键点
上传数据到 SRA 稍微麻烦点的就是填写项目信息表单了,先把数据通过 FTP 上传到 SRA 服务器,然后填好项目信息表单,就成功了。
数据属性信息格式
填写 attributes file 时候,每一列都按照文件表头的示例格式来,没有示例格式的可以自己根据实际项目信息填写。
表头绿色代表必须填写的项目(如果没有信息用 'not collected','not applicable','missing' 其中一个代替 ),蓝色代表至少需要填写一栏的项目(如果没有信息用 'not collected','not applicable','missing' 其中一个代替 ),黄色是可选项目(可以不填或者删除)。
填写好之后,另存为 TSV 格式文本文件才能上传,否则报错:无法识别文件编码。
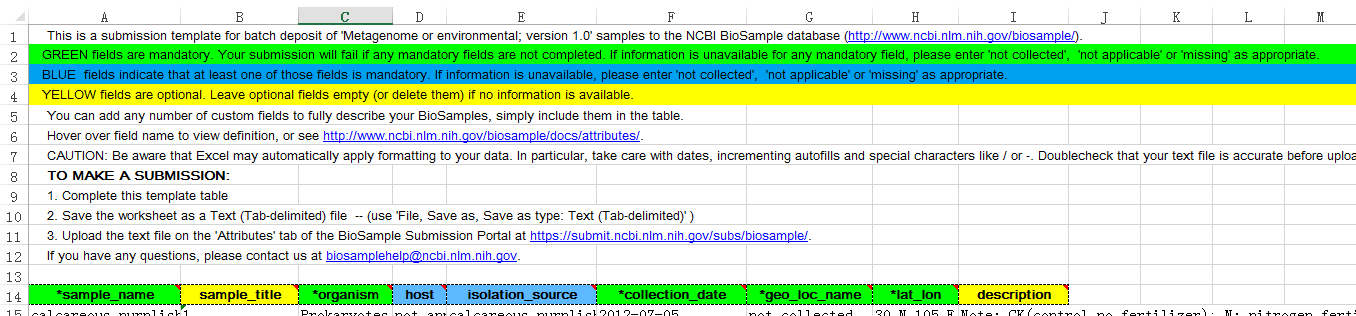 不同颜色 column 对应不同的要求
不同颜色 column 对应不同的要求
文件名格式
填写 SRA_metadata 的时候 sample_name 这一栏要与之前 attributes file 中天的 Biosample name (即上图中绿色的 *sample_name)一致,不是 Biosample 的编号,如果事先申请了 Bioproject 就填写,如果没有申请就空着。
特别注意文件名 filename,filename2,filename3...这些栏目,文件名与需要上传的文件完全相同(包括文件拓展名),例如上传文件 S1_1.fastq.gz,S1_2.fastq.gz 就填写文件名就是 filename:S1_1.fastq.gz,filename2:S1_2.fastq.gz。但是需要注意,如果是 tar 打包的文件则需要填写打包之前的原始文件名(包括拓展名),例如 S1_1.fastq.gz,S1_2.fastq.gz 被打包在一个文件 S1.tar 里面,文件名依然要分开填写 filename:S1_1.fastq.gz,filename2:S1_2.fastq.gz。
上传数据属性信息文件之前的格式检查
有些不用的 column 例如下图红色框中的,直接删除,防止报错。
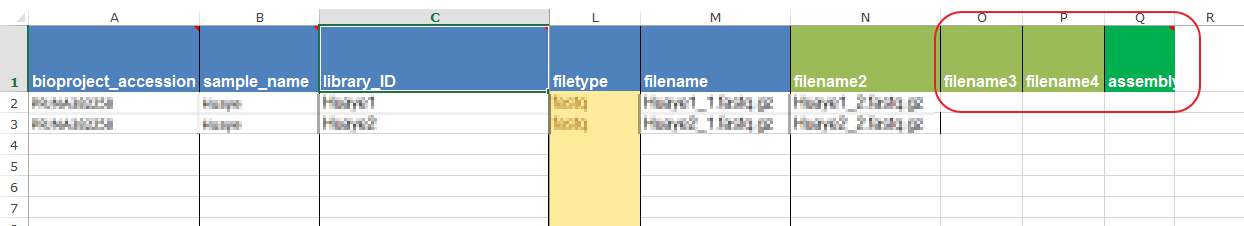 空出来的 column 可以删除
空出来的 column 可以删除
使用 Filezilla 连接 NCBI 进入个人目录
使用 filezilla 上传数据时,使用 SRA 提供的账号密码链接服务器后,状态提示栏会报 550/:permission denied 错误。此时只需要在软件的远程站点框中输入指定的个人目录 upload/xxxxx,然后 Enter 即可进入个人目录。
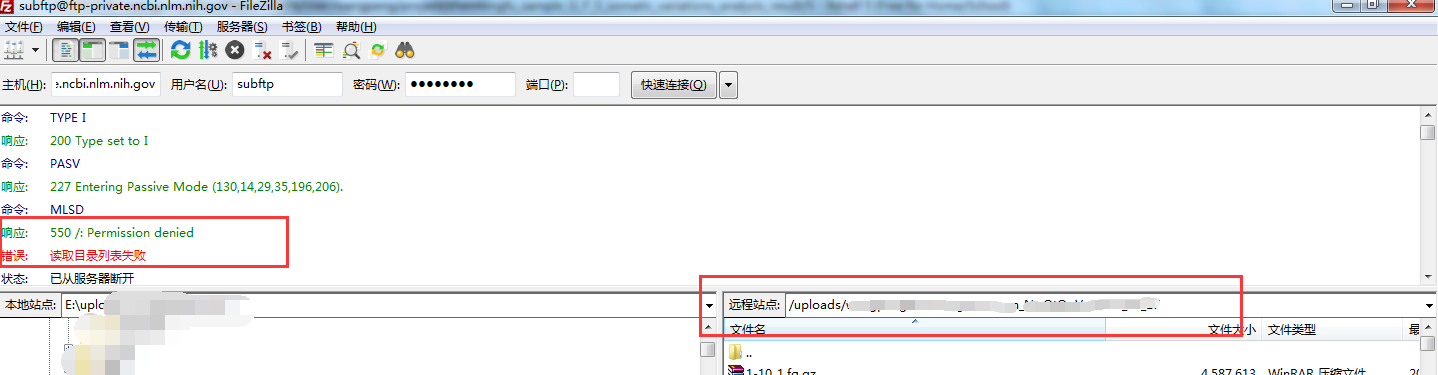 连接服务器后只能进入个人目录
连接服务器后只能进入个人目录
进入个人目录后需要新建一个文件夹,来上传数据。不能将数据直接上传到个人目录下面。
Filezilla 上传总是传输失败解决办法
在国内上传数据到 SRA 速度比较慢,网络也不是很稳定,可以通过如下设置减少传输失败的情况:
- 设置-传输-对已存文件的操作-上传:继续文件传输
- 设置-连接-超时-超时秒数:60s(默认的20s)
- 设置-连接-重连设置-最大重试次数:99次(调到最大)
- 设置-连接-重连设置-登录重试延时:90s(调大一点,防止网络不稳定时频繁重连可能被服务器拉黑)
最后更新:2017.5.7










网友评论